ESQUEMA GENERAL
Ø PROPIEDADES GENERALES DE LAS PROTEÍNAS Y AMINOÁCIDOS
· Aminoácidos
· Proteínas
Ø LA FORMA Y ESTRUCTURA DE LAS PROTEÍNAS.
· La forma de una proteína es especifica por su secuencia de aminoácidos
· Las proteínas se pliegan en una conformación de menor energía
· Las proteínas adoptan una gran variedad de formas complejas
· La hélice α y la lámina β son patrones de plegamiento comunes
· Las proteínas tienen varios niveles de organización
· Enlace peptídico
· Conformaciones peptídicas
Ø FUNCIONES GENERALES DE LAS PROTEÍNAS.
· Cómo funcionan las proteínas
· Todas las proteínas se unen a otras moléculas
· Las enzimas son catalizadores potentes y muy específicos
· Algunos ejemplos de funciones generales de las proteínas
Ø SÍNTESIS DE PROTEÍNAS.
· ARN de transferencia
· La fidelidad de la síntesis depende de los AA-ARNt sintetasas
· Ribosomas y síntesis de proteínas
Ø ÍNDICE ANALÍTICO
·
Cuando se examina una célula al microscopio o se analiza su actividad eléctrica o bioquímica, esencialmente se están observando proteínas. Las proteínas constituyen las unidades estructurales a partir de las cuales se ensamblan las células, y representan la mayor parte de su masa seca. Pero, además de proporcionarle forma y estructura a la célula, las proteínas también llevan a cabo la mayor parte de sus numerosas funciones. La multiplicidad de funciones que desempeñan las proteínas son el resultado de gran numero de formas diferentes que adoptan: la estructura dicta la función. De nodo tal empezaremos primero con el análisis de su estructura tridimensional. A continuación s comentará cómo funcionan las proteínas. Más tarde examinaremos como se regula la actividad proteica en las células y cómo afectan su función los cambios en la estructura de una proteína. Por último entraremos en la síntesis de proteínas y analizaremos de que manera convergen todos los componentes involucrados en la síntesis convergen ara realizar la última etapa en el flujo de la información genética, la traducción.
1. PROPIEDADES GENERALES DE LAS PROTEÍNAS Y AMINOÁCIDOS
· Aminoácidos.
Los átomos del carbono de los aminoácidos, a excepción de la glicocola[1], están unidos a cuatro grupos químicos diferentes. Esto es lo que caracteriza a un átomo de carbono asimétrico.Considerando la fórmula general de los aminoácidos y su relación en el espacio con las valencias dispuestas tetraédricamente del átomo de carbono asimétrico, los isómeros de una molécula pueden ser representados por no más de dos modelos tridimensionales (fig. 1). Ambos modelos son imágenes espectaculares uno de otro si el grupo R es idéntico en uno y en otro modelo y si por sí mismo no contiene otros centros de asimetría, siendo cada isómero óptimamente activo. Los dos isómeros difieren solamente en la dirección en que desvían el plano de la luz polarizada.
|
Espejo
COOH COOH | | H N --- C --- H H --- C --- NH | | R R |
Fig.1.Modelos generales de los enantiomorfos -amonoácidos.
Estos pares isómeros se denominan enantiomorfos. Un compuesto que hace girar la luz polarizada en el sentido de las agujas del reloj se denomina dextrorrotatorio (+) en oposición al compuesto que es levorrotatorio (-). [ I ].
· Proteínas.
Las proteínas son biopolímeros formados por unidades monoméricas llamadas -aminoácidos. Existen veinte -aminoácidos de importancia biológica. El enlace polimérico es formado por medio de la interacción del grupo amino de un aminoácido con el grupo carboxilo de otro aminoácido hasta formar el enlace peptídico. De este modo dos aminoácidos condensados forman un dipéptido.
La ulterior adición secuencial de residuos aminoácidos da lugar a un polipéptido y finalmente a una proteína.
La secuencia de los aminoácidos en cada proteína es única y de alguna manera determina la estructura general de la molécula. A la estructura particular o conformación de las proteínas en las células vivas se le llama estado natural ,las cuales no son fácilmente digeribles. Por esta razón las proteínas de la dieta son desnaturalizadas por medio de la cocción, la trituración o las transformaciones quimicas. Las estructuras naturales son destruidas en estos procesos para que las enzimas del sistema digestivo puedan hidrolizar mas rapidamente los enlaces peptídicos y de este modo liberar los componentes aminoácidos. Esta reacción da comienzo en el estómago por medio de las enzimas pepsina y renina en un medio acido de ph 1 y se completa en el intestino delgado. El jugo pancreático tambien contiene enzimas proteolíticas y peptidolíticas. Las aminopeptidasas y dipeptidasas de las células de la mucosa intestinal completan la hidrólisis de los peptidos resultantes para dar aminoácidos libres ,los cuales son transportados al sistema portal y desde allí a las células del cuerpo que los usaran según las necesidades de la homeostasis. [ I ].
2. LA FORMA Y LA ESTRUCTURA DE LAS PROTEINAS
Las proteínas son las moléculas conocidas de mayor complejidad estructural y refinamiento funcional. La estructura y la actividad de cada proteína ha sido desarrollada y puesta a punto durante millones de años de historia evolutiva.
La forma de una proteína es especificada por su secuencia de aminoácidos.
Las proteínas, se ensamblan a partir de un grupo de 20 aminoácidos, cada uno con distintas propiedades químicas. Una molécula de proteína se elabora a partir de una larga cadena de esos aminoácidos, cada uno ligado a su vecino por un enlace peptídico covalente. Las proteínas, por esta razón, también se denominan polipéptidos. Cada tipo de proteína tiene una secuencia particular de aminoácidos, exactamente la misma de una molécula a otra del mismo tipo.
Cada cadena polipeptídica consta de un esqueleto que sostiene a las distintas cadenas laterales de aminoácidos. El esqueleto polipeptídico se forma a partir de una secuencia repetida de átomos a lo largo de la cadena. Unida a esta cadena repetitiva se encuentra cualquiera de las cadenas laterales de los 20 aminoácidos,es decir, las partes de los aminoácidos que no intervienen en la formación del enlace peptídico.Algunos son no polares e hidrófobos, otros tienen carga negativa o positiva, otros son químicamente reactivos, etc.
Las cadenas largas de polipéptidos son muy flexibles: muchos de los enlaces covalentes que unen átomos de carbono en una cadena extensa de aminoácidos permiten la rotación libre de los átomos que unen. Por lo tanto, las proteínas en principio pueden plegarse de muchas formas.
Los enlaces no covalentes que ayudan a mantener la forma de las proteínas son los enlaces de hidrógeno, los enlaces iónicos y las fuerzas de van der Waals[2]. Puesto que los enlaces no covalentes son mucho más débiles que los enlaces covalentes, se necesitan muchos no covalentes para mantener unidas a dos regiones de una cadena polipeptídica.
Las moléculas hidrófobas pueden ser forzadas a unirse en un ambiente acuoso mediante la reducción de sus efectos de separación sobre la red de enlaces de hidrógeno de las moléculas de agua que las rodean. Por lo tanto un factor importante que gobierna el plegamiento de cualquier proteína es la distribución de sus aminoácidos polares y no polares .Las cadenas laterales no polares tienden a agruparse en el interior de una proteína plegada pudiendo evitar el contacto con el citosol acuoso que las rodea dentro de la célula. En contraste, las cadenas laterales polares, tienden por sí mismas a disponerse cerca del exterior de la proteína plegada,donde pueden formar enlaces de hidrógeno con el agua y con otras moléculas polares.
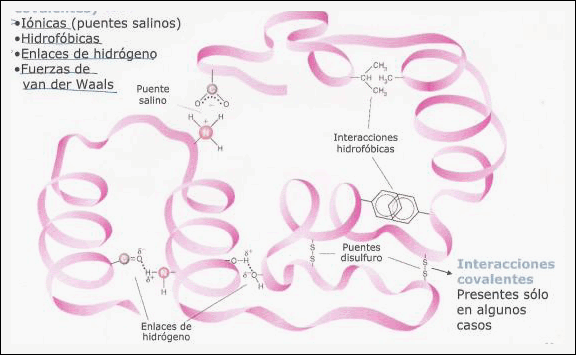
Fig.2. Fuerzas operativas en el plegamiento de las proteínas.
Las proteínas se pliegan en una conformación de menor energía.
La estructura plegada final, o conformación, que adopta cualquier cadena polipeptídica es regida por aspectos energéticos: una proteína generalmente se pliega en la forma en la cual la energía libre disminuye.
Cada proteína se pliega normalmente en una única conformación estable. Sin embargo, esta conformación a menudo cambia ligeramente cuando la proteína interactúa con otras moléculas en la célula. Ese cambio en la forma es crucial para la función de la proteína.
Cuando las proteínas se pliegan inapropiadamente pueden formar agregados susceptibles de dañar a las células e incluso a tejidos enteros. El agregado de proteínas es la base de diversas afecciones neurodegenerativas, como la enfermedad de Alzheimer y la enfermedad de Huntington[3].
Aunque una cadena proteica puede plegarse en su conformación correcta sin ayuda externa, el plegamiento de una proteína en una célula viva generalmente es asistido por proteínas especiales denominadas moléculas chaperonas. Estas proteínas se unen a cadenas parcialmente plegadas y las ayudan a plegarse a lo largo de la vía energéticamente más favorable.
Las proteinas adoptan una gran variedad de formas complejas
Las proteínas pueden ser globulares o fibrosas; pueden formar filamentos, láminas, anillos o esferas.
La resolución de la estructura de una proteína a menudo comienza con la determinación de su secuencia de aminoácidos. En primer lugar, las células se rompen y sus proteínas se separan y purifican. El orden preciso de los aminoácidos en una proteína pura puede establecerse después de diferentes modos. Actualmente para determinar el orden de los aminoácidos en una proteína mediante la secuenciación del gen que la codifica. Hasta ahora, el único camino para descubrir el patrón de plegamiento preciso de cualquier proteína es por experimentación, mediante los métodos de rayos X o de resonancia magnética.
La hélice α y la lámina β son patrones de plegamiento comunes.
Se pueden hallar dos patrones de plegamiento regular en partes de ella. El primer patrón de plegamiento descubierto, denominado hélice α, se halló en la proteína α-queratina, que es abundante en la piel y sus anexos, como el pelo, las uñas y los cuernos. En el mismo año se encontró una segunda estructura plegada, denominada lámina β que se encontró en la proteína fibroína, el principal constituyente de la seda.
Estos dos patrones de plegamiento son particularmente comunes debido a que resultan de los enlaces de hidrógeno que se forman entre los grupos N-H y C=O en el esqueleto polipeptídico. Debido a que las cadenas laterales de aminoácidos no están involucradas en la formación de estos enlaces de hidrógeno, las hélices alfa y las láminas β pueden generarse por muchas secuencias diferentes de aminoácidos.
Las proteínas tienen varios niveles de organización.
La estructura de una proteina comienza con su secuencia de aminoácidos, que por ello es considerada su estructura primaria. El nivel de organización siguiente abarca a las hélices alfa y a las láminas beta que se forman dentro de ciertos segmentos de una cadena polipeptídica; estos plieges son los elementos de la estructura secundaria de la proteína. La conformación tridimensional completa formada por una cadena polipeptídica entera, que comprende las hélices alfa, las láminas beta, los enrollamientos al azar y cualquier otra asa y pliege que se forme entre el N-terminal y el C-terminal, se denomina a veces estructura terciaria. Finalmente, si una molécula proteica particular se forma como complejo de más de una cadena polipeptídica la estructura completa se designa estructura cuaternaria.[ II ].
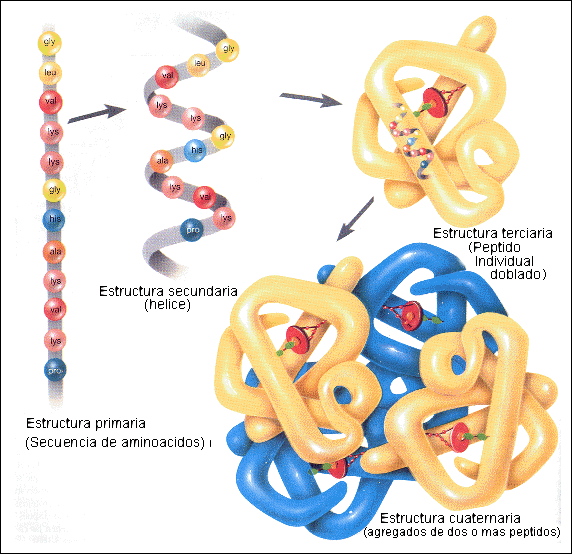
Fig.3. Los 4 niveles estructurales de las proteinas.
Enlace peptídico
Un polipéptido se forma como resultado de la condensación de aminoácidos, produciéndose una molécula lineal. Un extremo de esta hilera de residuos de aminoácidos tiene un grupo amino libre, y en el otro extremo existe un grupo carboxilo libre. Los polipéptidos se denominan como derivados del aminoácido con el grupo carboxilo libre.
La secuencia peptídica se escribe con el –NH3+ terminal a la izquierda. Cada aminoácido puede ser abreviado mediante un símbolo de tres letras de su nombre en inglés.
No existe una clara diferenciación entre polipéptido y proteína; el término “proteína” se refiere normalmente a un biopolímero con peso molecular superior a varios miles. Un polipéptido tiene un peso molecular bajo, y debe indicarse el número de aminoácidos de la molécula si se conoce.
Conformaciones peptídicas
La condensación del grupo amino de un aminoácido con el grupo carboxilo de otro forma un enlace peptídico que posee una estabilización de resonancia que resulta por una disposición planar de los seis átomos implicados. La conformación constituye la configuración tridimensional de la molécula. Otras propiedades del polipéptido tienden a estabilizar la molécula a una forma más rígida.
Los elementos estabilizadores de la estructura son puentes de hidrógeno, interacciones iónicas e interacciones hidrofóbicas. La contribución a la estabilidad de cada interacción iónica en la conformación de una molécula depende en gran parte de la separación entre los grupos, y puede ser mayor que la estabilización resultante de un enlace de hidrógeno sencillo. [ I ].
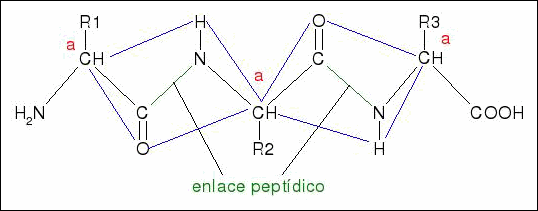
Fig. 4. Conformación del enlace peptíco.
3. FUNCIONES GENERALES DE LAS PROTEÍNAS
Cómo funcionan las proteínas.
Es la conformación de la proteína la que otorga a ésta una función única basada en sus propiedades químicas y el preciso engranaje de sus partes cuyas acciones están acopladas a los procesos químicos.
Para las proteínas, por lo tanto, forma y función están inexorablemente ligadas.
Todas las proteínas se unen a otras moléculas
Las propiedades biológicas de una molécula proteica dependen de su interacción física con otras moléculas. Los anticuerpos se unen a los virus o bacterias como una señal para las defensas del cuerpo, la enzima hexocinasa une glucosa y ATP para catalizar una reacción entre éstos, las moléculas de actina se unen entre sí para ensamblarse en largos filamentos, etc.
Todas las proteínas se adhieren, o unen, a otras moléculas. En algunos casos esta unión es muy fuerte; en otras ocasiones es débil y de corto plazo. La unión muestra siempre gran especificidad ya que cada molécula proteica puede unirse solo a una o a unas pocas moléculas entre muchos miles de diferentes moléculas con las que se encuentra. Cualquier sustancia que es unida por una proteína se denomina ligando de esta proteína.
Las enzimas son catalizadores potentes y muy específicos.
Para muchas proteínas, la unión a otra molécula es su única función. Una molécula de anticuerpo sólo necesita unirse a su molécula diana sobre la superficie de una bacteria o un virus y su trabajo está cumplido; una molécula de actina solo necesita asociarse con otras moléculas de actina para formar un filamento. Sin embargo, hay otras proteínas para las cuales la unión al ligando es simplemente una primera etapa necesaria en su función. Esta es el caso de las enzimas, las cuales determinan todas las transformaciones químicas que se producen en las células. Las enzimas se unen a uno o más ligandos, denominados sustratos, y los convierten en productos modificados químicamente, y hace esto una y otra vez con asombrosa rapidez. [ II ].
 Algunos ejemplos de funciones generales de las proteínas
Algunos ejemplos de funciones generales de las proteínas
ENZIMA.
Función: catálisis de la ruptura o formación de un enlace covalente.
Ejemplos: triptófano sintetasa, elabora al aminoácido triptófano.
PROTEINAS ESTRUCTURALES.
Función: proporcionar soporte mecánico a las células y tejidos.
Ejemplos: colágeno y elastina.
PROTEINAS DE TRANSPORTE.
Función: transportar moléculas pequeñas o iones.
Ejemplos: la albúmina y la hemoglobina.
PROTEINAS MOTORAS.
Función: generar movimientos en las células y tejidos.
Ejemplos: la miosina.
PROTEINAS DE DEPOSITO.
Función: almacenar pequeñas moléculas o iones.
Ejemplo: el hierrro se almacena en el hígado.
PROTEINAS DE SEÑALIZACIÓN.
Función: transmitir señales de una célula a la otra.
Ejemplo: hormonas y factores de crecimiento.
PROTEINAS RECEPTORAS.
Función: detectar señales y transmitirlas a la maquinaria de respuesta de la célula.
Ejemplo: la rodopsina en la retina detecta la luz.
PROTEINAS REGULADORAS GÉNICAS.
Función: unirse al DNA para activar o desactivar genes.
Ejemplo: el represor de la lactosa en las bacterias silencia a los genes para las enzimas que degradan al azúcar lactosa.
PROTEINAS ESPECIALES.
Función: muy variable.
Ejemplo: los organismos producen muchas proteínas con propiedades muy especializadas. Las proteínas anticongelantes de los peces del Ártico y de la Ántartida protegen a su sangre contra el congelamiento. [ II ].
Fig.5. Triple hélice de tropocolágeno.
4. SINTESIS DE PROTEINAS.
ARN de transferencia.
Los ARNs de transferencia constituyen una familia de ácidos ribonucleicos pequeños, que de cierta manera actúan como intérpretes del código genético. Los ARN ts son capaces de leer el mensaje expresado como codones en el ARNm, y al mismo tiempo pueden reconocerse a los aminoácidos especificados por estos codones. Se comprende fácilmente la necesidad de una molécula de ARN de trasferencia, ya que no existe una afinidad específica entre los grupos laterales de los aminoácidos y las bases de los ARNm. Este paso fundamental de la traducción es realizado por enzimas activantes, las AA-ARNt sintetasas,que unen a los aminoácidos a un extremo de la molécula del ARNt, y por tripletes específicos de bases (anticodones) capaces de ligarse a los codones correspondientes del ARNm mediante uniones de hidrógeno. Por cada uno de los 20 amoniácidos debe existir por lo menos uno o más ARNt específicos.
Los ARNt de transferencia se asemejan en los extremos de su cadena, tanto en el 3´, que siempre termina en CCA y se une al aminoácido específico, como en el 5´, que frecuentemente termina con una guanina. En todos los ARNs de transferencia se encuentra una secuencia en una región especial de la molécula, el sitio para el reconocimiento del ribosoma.
La característica principal del modelo en hoja de trébol es la existencia de una serie de asas separadas por ejes cortos, formados por regiones de 4 a 7 bases complementarias, donde el ARN es bicatenario y se dispone en hélice. Los principales elementos del modelo son:
1) el extremo aceptor ( CCA ) en 3´donde se une al aminoácido;
2) el anticodón, un asa con tres bases que reconocer y forman puentes de hidrógeno con el codón en el ARNm, lo que permite leer el mensaje genético;
3) el asa D, que sirve para el reconocimieto de la aminoacil-ARNt-sintetasa;
4) El asa T, que interactúa con una zona complementaria del ARNr de 5 S durante la síntesis de proteína.[III]
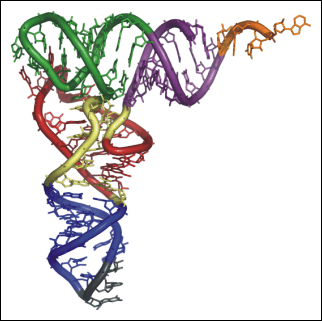
Fig.6. Estructura del ARN de transferencia.
La fidelidad de la síntesis depende de las AA-ARNt sintetasas.
La función del ARNt es esencial en dos pasos que determinan la fidelidad de la síntesis de la proteína. En primer lugar, selecciona el aminoácido correcto, que se le unirá para formar el aminoacil-ARNt; luego selecciona el codón correspondiente en el ARNm.
Los aminoácidos se unen a los ARNts por medio de enzimas específicas, las AA-ARNt sintetasas o enzimas activantes a los AA. La exactitud de la síntesis de proteína depende de que la sintetasa una el ARNt con el AA correcto, ya que el ARNm solo es capaz de reconocer el ARNt. Por lo tanto, las ARNt sintetasas realizan una etapa crucial en la traducción del código genético.
El AA-AMP que se forma como paso intermedio permanece unido a la enzima hasta que llega el ARNt adecuado, produciéndose en ese momento la formación del complejo AA- ARNt.
La enzima activante tiene dos sitios: uno de reconocimiento del aminoácido y otro de reconocimiento del ARNt específico.
La unión entre el aminoácido y el extremo CCA del ARNt es de alta energía; ésta proviene del ATP usado en la reacción y será luego usada para impulsar la formación de la unión peptídico en la síntesis.
La extraordinaria exactitud de las ARNt sintetasas se debe a que es una reacción de dos etapas. El proceso llamado de lectura ( Prof. Reading), es esencial para evitar equivocaciones en la lectura del código genético.[III]
RIBOSOMAS Y SÍNTESIS DE PROTEINAS.
Los ribosomas son estructuras macromoleculares que guían las múltiples interacciones involucradas en la síntesis de proteínas. Para alcanzar éste objetivo, los ribosomas funcionan en grupos denominados polirribosomas o polisomas. El número de ribosomas en un polisoma está relacionado con la longitud de la proteína que debe ser sintetizada; por ejemplo, para los polipéptidos de la hemoglobina se emplea un pentámero ( 5 ribosomas). Cada ribosoma lleva una cadena polipéptidica naciente, es decir, que en el caso de la hemoglobina se forman simultáneamente cinco cadenas a lo largo del ARNm.
Los polisomas junto con el ARNm, pueden ser extendidos y observados con el microscopio electrónico. El proceso de trascripción de las bacterias es simultáneo con el de traducción, es decir, que los ribosomas se unen al ARNm mientras todavía ocurre la trascripción de éste. Con el microscopio electrónico también pueden observarse los puntos de contacto de la polimerasa del ARN con el molde de ADN.
En los ribosomas libres de bacterias y eucariontes puede demostrarse la existencia de un ciclo ribosoma-polisoma. Ambas subunidades ribosómicas se unen en la iniciación y se disocian en la terminación. De esta manera se intercambian las subunidades y existe una población de subunidades libres. En la separación de las subunidades al finalizar la traducción interviene un factor de disociación de naturaleza proteica, idéntica al factor de iniciación IF3.
Las 2 subunidades ribosómicas ejecutan distintas funciones. La 30S con el factor IF3 se une al ARNm e el sitio del ribosoma, que incluye al codón AUG. En las bacterias el alineamiento correcto para el comienzo de la síntesis depende de un a secuencia de 3 a 8 bases en este sitio de unión del ribosoma, bases que son complementarias de otras en el extremo 3´ del ARN de 16S. En el complejo de iniciación también pueden intervenir asas-horquilla de ARNm.
En las bacterias del codón AUG inicial codifica a la formilmetionina, y en las eucariontes, a la metionina. Estos aminoácidos iniciales son luego eliminados por enzimas. Existen 3 factores proteicos, IF1, IF2 e IF3, laxamente asociados, con la subunidad ribosómica pequeña que participan en la iniciación de síntesis de proteinas. IF1 + IF2, junto con GTP, son requeridos para unir el F-met-ARNt a la subunidad pequeña en respuesta al codón[4] AUG. El factor IG3 estimula la unión del ARNm al sitio de iniciación de la subunidad pequeña. Este complejo de iniciación de la subunidad 40S con el IF3 puede ser estudiado al microscopio electrónico.
En las eucariontes también se han separado varios factores de inicición y se ha encontrado que en la fosforilación del IF2, que es regulada por el AMPc, podría ser importante en el control de la velocidad de la síntesis de proteína.
Los factores proteicos empleados en el alargamiento de la cadena polipeptídica se encuentran en el sobrenadante posribosómico. El factor EFT comprende las proteínas Tu y Ts. EFTu + GTP forman un complejo con el aminoacil-ARNt, y la formación de este complejo es catalizada por el factor EFTs. En contacto con el ribosoma, el factor EFG, denominado traslocasa, desdobla al GTP en GDP + Pi. La energía liberada se emplea en la expulsión del ARNt vacío y en proceso de traslocación.
La síntesis de proteína consume mucha energía; para la incorporación de un aminoácido se usa un ATP y dos GTPs.
La subunidad ribosómica mayor contiene la enzima peptidiltransferasa, que interviene en la formación de la unión peptídica. Esta subunidad también posee 2 sitios de unión: el sitio aminoácido o aceptor, y el sitio peptidílico o dador. La subunidad mayor se agrega después del enganche del F-met-ARNt con el codón AUG sobre la subunidad pequeña. El crecimiento da la cadena polipeptídica involucra: a) la entrada de una aminoacil-ARNt al sitio aminoacídico; b) la formación de una unión peptídica y la liberación del ARNt vacío que ocupaba el sitio peptídico; c) el movimiento del peptidil-ARNt desde el sitio aminoacídico hacia el peptídico; d) la entrada de un nuevo aminoacil-ARNt en el sitio aminoacídico. Simultáneamente, al ARNm se mueve en la dirección 5´- 3´ para presentar el codón siguiente. Todos estos pasos coordinados se realizan a gran velocidad ( una cadena de hemoglobina de 150 aminoácidos por minuto). La terminación de la cadena polipeptídica ocurre cuando se alcanza un codón de terminación ( UAG, UAA, UGA). En este proceso participan 2 factores proteicos de terminación, R1 y R2.
Los antibióticos intervienen con etapas precisas de la síntesis de proteínas y ésta es la base molecular de su uso en medicina. Otras drogas actúan sobre la síntesis del ARN, como actinomicina y a- amanitina.
En las eucariontes las proteínas de membrana y las secretorias se originan en los ribosomas unidos a membrana de RE. Las proteínas exportadas tienen un péptido señal hidrofóbico, que es codificado por los codones iniciales del ARNm. Este péptido señal luego se elimina por acción de una peptidasa específica. [III] .
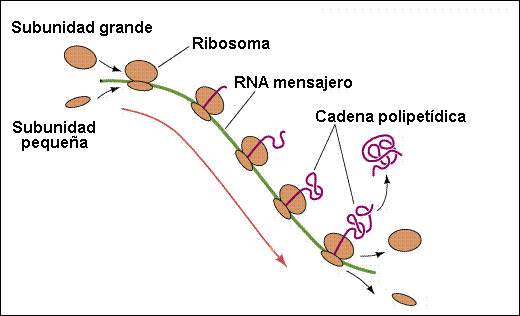
Fig. 7. Esquema de la síntesis de proteínas.
Fig.1. Montgomery, R et al, 1980, Bioquímica médica. Barcelona: Salvat Editores, S.A.
Fig.2. Alberts, B. et al, 2006, Introducción a la Biología Celular. Madrid: editorial médica Panamericana S.A.
Fig.3. Raisman, J.S; González A.M, Hipertextos del área de la Biología (en línea). UNNE. Disponible en: http://www.biologia.edu.ar/macromoléculas/structup.htm Visitado en:12/05/07.
Fig.4. Wikipedia “la enciclopedia libre” (en línea). Disponible en: http://es.wikipedia.org/wiki/enlace_pept%C3%Addico. Visitado en: 26/06/07.
Fig.5. Wikipedia “la enciclopedia libre” (en línea). Disponible en: http://es.wikipedia.org/wiki/Hélice_de_colágeno. Visitado en: 30/06/07.
Fig.6. Wikipedia “la enciclopedia libre” (en línea). Disponible en: http://es.wikipedia.org/wiki/ARN_de_transferencia.
Fig. 7. Raisman, J.S; González A.M. Hipertextos del área de la Biología (en línea). UNNE. Disponible en: http://www.efn.uncor.edu/dep/biologia/intrbiol/adntema2.htm Visitada en: 30/05/07.
Alberts, B. et al, 2006, Introducción a la Biología Celular. Madrid: editorial médica Panamericana S.A. [II].
De Robertis,E; De Robertis, E (h); 1981, Biología celular y molecular. Barcelona: “El Ateneo”. [III].
Montgomery, R et al, 1980, Bioquímica médica. Barcelona: Salvat Editores, S.A. [I].
[1] Aminoácido proteico más sencillo. También se denomina glicina y antiguamente se denominó azúcar de gelatina.
[2] Johannes Diderik van der Waals. Fue el primero en darse cuenta de la necesidad de tomar en consideración el volumen de la s moléculas y las fuerzas intermoleculares (fuerzas de Van der Waals), estableciendo la relación entre presión, volumen y temperatura de los gases y de los líquidos.
[3] Enfermedad neurológica, degenerativa, autosómica y dominante, que afecta al cerebro dónde las neuronas van degenerándose y finalmente mueren.
[4] La información genética, contenida en el ARNm, se escribe a partir de 4 letras que corresponden a las bases nitrogenadas ( A, C, G, T) , las cuales van agrupadas de tres en tres. Cada grupo de tres se llama codón y lo que hace es codificar un aminoácido o un símbolo de puntuación ( comienzo, parada).
 o vende lo que ya no usas
o vende lo que ya no usas